I. GIỚI THIỆU
Cây lanh (Linum usitatissimum L.) là một loại cây trồng đa mục đích được chia thành cây lanh sợi, cây lanh dầu và cây lanh kép lấy sợi dầu (Xie et al., 2018). Sợi lanh có độ bền kéo cao, mềm mại, hấp thụ và phân tán nước nhanh, độ giãn nở cao, có thể kéo thành sợi có số lượng lớn, được sử dụng trong sản xuất quần áo cao cấp và thay thế cho sợi thủy tinh (Mahboob et al., 2017). Cây lanh đã được sử dụng trong các lĩnh vực vật liệu tổng hợp sinh học, y học và các lĩnh vực khác, do đó làm tăng giá trị kinh tế của nó (Barkoula, Garkhail, Peijs, & Products, 2010; Prasad, 2000). Hạt lanh là một loại hạt có dầu được sử dụng trong các sản phẩm sức khỏe tự nhiên. Các thành phần dinh dưỡng được tìm thấy trong hạt lanh bao gồm lignan, axit linolenic, axit linoleic, lipid, protein, chất xơ, carbs và vi chất dinh dưỡng (Shim et al., 2014). Protein hạt lanh đã được áp dụng cho nhiều loại sản phẩm thực phẩm chức năng, như sữa, sản phẩm bánh mì và sản phẩm thịt (Goyal, Sharma, Upadhyay, Gill, & Sihag, 2014). Dầu hạt lanh chứa axit béo không bão hòa đa, có tác dụng ngăn ngừa tình trạng tăng lipid máu và xơ vữa động mạch.
Ở thực vật, nhân tố phiên mã (transcription factor - TF) đóng vai trò trung tâm trong điều hòa các phản ứng sinh lý diễn ra trong suốt quá trình sinh trưởng và phát triển (Liu et al., 2023). Thông qua việc bám trên vùng promoter, TF giúp điều hòa (kìm hãm hoặc thúc đẩy) sự biểu hiện của gene mục tiêu thông qua con đường tín hiệu phytohormone, giúp tế bào trả lời lại kích thích từ môi trường. Trong đó, cánh tay kẽm (zinc finger) là một trong những họ protein chức năng đặc trưng cho thực vật, gồm nhiều nhóm TF quan trọng, như nhân tố cảm ứng ethylene (ethylene responsive factor), nhân tố có vùng bảo thủ đặc trưng homeobox thực vật (plant homeobox domain), nhân tố giàu AT và mang vùng bảo thủ đặc trưng cánh tay kẽm (plant AT-rich sequences and zinc-binding, PLATZ). Các nghiên cứu gần đây đã ghi nhận vai trò của nhóm TF PLATZ tham gia vào các quá trình sinh học quan trọng trên các loài thực vật. Ví dụ, gene ZmPLATZ12, một thành viên trong nhóm TF PLATZ ở cây ngô (Zea mays), có biểu hiện đặc thù ở tế bào nội nhũ chứa tinh bột, được chứng minh là kiểm soát quá trình phát triển nội nhũ thông qua tương tác với phức hệ RNA polymerase III (J. Wang, Ji, Li, Zhou, & Wu, 2018). Một gene mã hóa TF PLATZ khác trên cây mô hình hai lá mầm Arabidopsis thaliana được ghi nhận tham gia vào cơ chế thúc đẩy sự biểu hiện của các gene liên quan đến kích thước và sự úa vàng ở lá (Kim et al., 2018). Gần đây, nhóm TF PLATZ đã được nghiên cứu trên một số đối tượng cây trồng, như Arabidopsis (González-Morales et al., 2016), ngô (J. Wang et al., 2018), lúa gạo (Oryza sativa) (A. Wang et al., 2019), lúa mỳ (Triticum aestivum) (Fu et al., 2020)cải bắp Trung Quốc (Brassica rapa) (Azim, Khan, Hassan, Robin, & biotechnology, 2020), các loài thuộc họ Hoa hồng (Rosaceae), bao gồm táo tây (Malus domestica) thuộc giống Golden delicious (Jiuyang Li et al., 2023), táo tây thuộc giống Hanfu (Sun et al., 2023), táo tây thuộc giống Gala (Sun et al., 2023), táo dại Tân Cương (M. sieversii) (Sun et al., 2023), táo dại Siberi (M. baccata) (Sun et al., 2023), táo dại châu Âu (M. sylvestris) (Sun et al., 2023), dâu tây dại (Fragaria vesca) (Sun et al., 2023), đào (Prunus persica) (Sun et al., 2023).
Trong nghiên cứu này, các gen LuPLATZ (Linum usitatissimum PLATZ) đã được xác định và định vị, đồng thời thực hiện phân tích phát sinh loài. Cấu trúc gen và tính liên kết giữa các đặc hiệu và giữa các gen của các gen LuPLATZ đã được nghiên cứu. Nghiên cứu này cung cấp nền tảng cho nghiên cứu bổ sung về vai trò sinh học của gen LuPLATZ trên cây lanh.
II. KẾT QUẢ
1. Xác định các thành viên họ gen LuPLATZ
Bộ gen cây lanh có 28 gen LuPLATZ , trong đó LuPLATZ18 mã hóa cho protein ngắn nhất (164 aa) và LuPLATZ21 mã hóa cho protein dài nhất (346 aa). Khối lượng phân tử của protein LuPLATZ là 17,67 (LuPLATZ18) đến 38,13 KDa (LuPLATZ21). Các protein cơ bản được mã hóa bởi gen LuPLATZ (pI > 7), ngoại trừ gen LuPLATZ4 mã hóa protein có tính axit (pI < 7). Hệ số không ổn định dao động từ 47,40 (LuPLATZ16) đến 83,00 (LuPLATZ25) và hệ số chất béo dao động từ 57,50 (LuPLATZ20) đến 77,20 (LuPLATZ18). Điểm GRAVY thay đổi từ −231 và −770. Phạm vi này phản ánh tính ưa nước đáng kể của protein LuPLATZ (Bảng 1)
Bảng 1. Thông tin thành viên họ gen LuPLATZ
|
Tên định danh
|
Tên gen
|
Chiều dài (aa)
|
MW (kDa)
|
số Pi
|
mục lục
|
chỉ số béo
|
độ ưa nước
|
Vị trí tế bào
|
|
|
|
L.us.o.m. scaffold67.213
|
LuPLAZT1
|
326
|
36,36
|
8,51
|
51,66
|
65,21
|
−0,618
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold6.44
|
LuPLAZT2
|
264
|
29,76
|
8,97
|
53,31
|
69,73
|
−0,418
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold22.74
|
LuPLAZT3
|
254
|
28,55
|
8,87
|
60,64
|
76,02
|
−0,231
|
lục lạp. Nhân tế bào.
|
|
|
L.us.o.m. scaffold59.34
|
LuPLAZT4
|
253
|
28,4
|
6,33
|
60,66
|
69,01
|
−0,510
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold230.3
|
LuPLAZT5
|
237
|
25,49
|
9,71
|
50,78
|
69,58
|
−0,330
|
lục lạp.
|
|
|
L.us.o.m. scaffold117.141
|
LuPLAZT6
|
216
|
24.34
|
8,99
|
62,49
|
64,54
|
−0,500
|
lục lạp. Nhân tế bào.
|
|
|
L.us.o.m. scaffold242.21
|
LuPLAZT7
|
215
|
24.13
|
9,11
|
61,43
|
62,14
|
−0,502
|
lục lạp. Nhân tế bào.
|
|
|
L.us.o.m. scaffold92.48
|
LuPLAZT8
|
234
|
25,2
|
9,13
|
53,58
|
72,09
|
−0,238
|
lục lạp.
|
|
|
L.us.o.m. scaffold32.37
|
LuPLAZT9
|
243
|
27,51
|
8,89
|
54,12
|
64,12
|
−0,558
|
Nhân tế bào.
|
|
|
L.us.o.m.s caffold77.120
|
LuPLAZT10
|
285
|
31,57
|
9,46
|
59,51
|
67,82
|
−0,381
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold2.342
|
LuPLAZT11
|
245
|
27,83
|
9,31
|
55,2
|
6.04
|
−0,769
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold2.584
|
LuPLAZT12
|
245
|
27,21
|
9,12
|
57,91
|
67,27
|
−0,456
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold102.109
|
LuPLAZT13
|
277
|
30,92
|
9,59
|
57,96
|
66,9
|
−0,416
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold21.141
|
LuPLAZT14
|
314
|
36.03
|
8,38
|
48,88
|
67,61
|
−0,513
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold229.43
|
LuPLAZT15
|
263
|
29,99
|
8,95
|
53,83
|
72,21
|
−0,387
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold106.152
|
LuPLAZT16
|
337
|
37,74
|
7,83
|
47,4
|
69,14
|
−0,487
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold219.40
|
LuPLAZT17
|
262
|
28,98
|
8,39
|
68.03
|
58,55
|
−0,510
|
lục lạp.
|
|
|
L.us.o.m. scaffold262.39
|
LuPLAZT18
|
164
|
17,67
|
1,14
|
53,19
|
77,2
|
−0,382
|
lục lạp. Nhân tế bào.
|
|
|
L.us.o.m. scaffold253.61
|
LuPLAZT19
|
269
|
29,71
|
7,93
|
66,98
|
59,55
|
−0,503
|
lục lạp.
|
|
|
L.us.o.m.scaffold8.223
|
LuPLAZT20
|
244
|
27,75
|
9,30
|
53,37
|
57,5
|
−0,770
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold144.27
|
LuPLAZT21
|
346
|
38,13
|
9.03
|
61,71
|
67,6
|
−0,411
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold19.22
|
LuPLAZT22
|
277
|
31,79
|
8,44
|
49,75
|
63,61
|
−0,679
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold111.147
|
LuPLAZT23
|
248
|
28,33
|
9,16
|
57,19
|
66,01
|
−0,654
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold57.304
|
LuPLAZT24
|
253
|
28,36
|
8,36
|
58,99
|
68,62
|
−0,517
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold64.75
|
LuPLAZT25
|
228
|
25,52
|
8,52
|
83
|
6,26
|
−0,707
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold14.249
|
LuPLAZT26
|
249
|
27,9
|
8,37
|
59,6
|
75,98
|
−0,250
|
lục lạp. Nhân tế bào.
|
|
|
L.us.o.m. scaffold291.33
|
LuPLAZT27
|
296
|
33,8
|
9,24
|
63,76
|
61,25
|
−0,691
|
Nhân tế bào.
|
|
|
L.us.o.m. scaffold409.15
|
LuPLAZT28
|
237
|
26,83
|
9,35
|
48,58
|
59,54
|
−0,680
|
Nhân tế bào.
|
|
2. Định vị gen LuPLATZ trên nhiễm sắc thể
Trong số 28 gen LuPLATZ , 27 gen được phân bố trên 14 nhiễm sắc thể (Chr) và không có protein PLATZ nào được định vị trên Chr 10. LuPLATZ28 nằm trên vị trí (không thể tập hợp thành nhiễm sắc thể) và hầu hết các gen LuPLATZ đều nằm ở các Chr. Ở Chr 3 có 4, tiếp theo là Chr 5 (3) và Chr 11 (3). Theo trình tự nhiễm sắc thể và vị trí của các gen trên nhiễm sắc thể, các thành viên họ LuPLATZ được đặt tên là LuPLATZ1-LuPLATZ28 (Hình 1).
    |
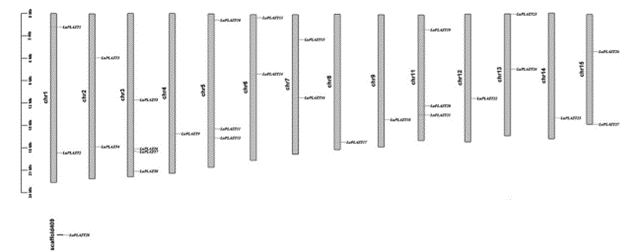 |
| Hình 1. Vị trí các gen LuPLATZ trên Chr |
3. Phân tích phát sinh loài
Để kiểm tra sự tiến hóa và mối quan hệ di truyền của protein LuPLATZ chi tiết hơn, chúng tôi đã chọn 181 chuỗi protein PLATZ từ cây lanh (28), Arabidopsis (12), cải dầu (24), cà chua (21), lúa mì (27), đậu nành ( 30), lúa (15), ngô (15), khoai tây (9) và đậu tương (30) để xây dựng cây phát sinh loài đa loài (Hình 2). 181 thành viên họ PLATZ từ các loài khác nhau được phân loại thành bảy phân họ (AG) dựa trên cấu trúc liên kết của cây phát sinh gen. Sáu nhóm protein LuPLATZ bao gồm phân họ E không có thành viên LuPLATZ và các nhóm A, B, C, D, F và G lần lượt có 6, 4, 3, 3, 3, 8 và 4 protein LuPLATZ. So với các cây một lá mầm là lúa, ngô và lúa mì, hầu hết các thành viên họ LuPLATZ có quan hệ gần gũi hơn với PLATZ của các cây hai lá mầm như đậu tương, A. thaliana , cà chua, khoai tây và cải dầu, cho thấy rằng LuPLATZ gần với một tổ tiên hai lá mầm hơn tổ tiên một lá mầm.
    |
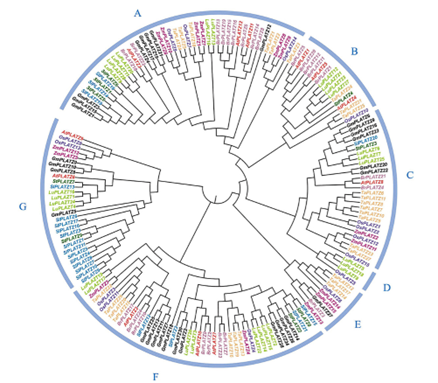 |
| Hình 2. Phân tích phát sinh loài của protein LuPLATZ bằng cách sử dụng protein PLATZ từ tám loài. Lưu ý: protein PLATZ ở mỗi loài được đánh dấu bằng một màu. Tên của mỗi protein PLATZ được đặt trước bởi hai chữ cái viết tắt của loài, cho biết nguồn gốc của nó. Lu cho cây lanh, at cho Arabidopsis, sl cho cà chua (Solanum lycopersicum), Ta cho lúa mì, Br cho cải bắp rapa L., st cho khoai tây (Solanum tuberosum), Os cho gạo, gm cho đậu tương và zm cho ngô. Cây phát sinh gen được chia thành các vùng A – G dựa trên mối quan hệ tiến hóa. |
4. Dự đoán các sự kiện nhân đôi các gen LuPLATZ
Phân tích có hệ thống về khả năng sao chép gen trong bộ gen cây lanh để hiểu rõ hơn về sự tiến hóa của gen LuPLATZ trong cây lanh. Phần mềm TBtools đã được sử dụng để thực hiện so sánh chi tiết toàn bộ bộ gen cây lanh dựa trên sự tương đồng về trình tự axit amin. Hai mươi bảy cặp sao chép đoạn đã được xác định trong gen LuPLATZ , với khả năng sao chép song song ở LuPLATZ6 và LuPLATZ7 nằm trên nhiễm sắc thể thứ ba (Hình 3). Các phát hiện cho thấy tần suất các sự kiện tái diễn có tác động lớn đến sự tiến hóa của gen LuPLATZ . Sử dụng cây lúa và cây Arabidopsis làm mô hình cộng sinh giữa các loài, chúng tôi đã suy ra lịch sử tiến hóa và vai trò chức năng của họ gen LuPLATZ (Hình 4). Phân tích tương đồng cho thấy chỉ có một gen OsPLATZ được liên kết với ba gen LuPLATZ ở cây lúa. Ở thực vật hai lá mầm, tám cặp gen tương đồng trực tiếp đã được xác định trong gen Arabidopsis AtPLATZ và gen LuPLATZ của cây lanh . LuPLATZ11 được ghép cặp tương đồng với ba gen AtPLATZ ở cây Arabidopsis, cho thấy vai trò tiềm năng của LuPLATZ11 trong sự phát triển của họ gen LuPLATZ . Điều quan trọng là nghiên cứu mối quan hệ giữa các loài và dự đoán chức năng gen.
    |
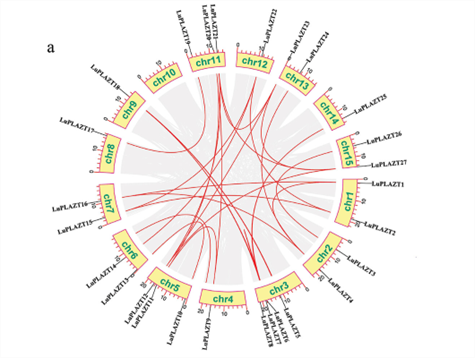 |
| Hình 3. Phân tích liên kết của gen LuPLATZ . Chiều dài của hộp nhiễm sắc thể biểu thị chiều dài thực tế của nó. Các gen được kết nối bằng các đường màu đỏ biểu thị sự sao chép gen một phần. |
    |
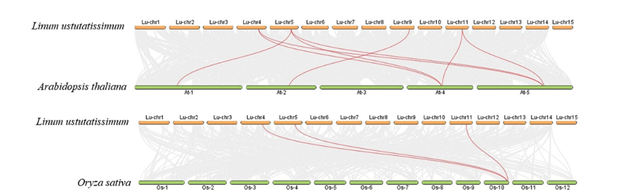 |
| Hình 4. Phân tích tính liên kết giữa các gen LuPLATZ với gen Arabidopsis thaliana và lúa PLATZ . Các cặp cây lanh tương đồng khác loài với cây Arabidopsis và cây lúa được nối với nhau bằng các đường màu đỏ. |
III. THẢO LUẬN VÀ KẾT LUẬN
PLATZ-TF là một loại protein liên kết DNA phụ thuộc kẽm, rất quan trọng cho sự phát triển của hạt (Han et al., 2022). 28 gen LuPLATZ đã được xác định trong nghiên cứu về bộ gen cây lanh này và được đặt tên là LuPLATZ1 – LuPLATZ28 . Số lượng họ gen PLATZ ở cây lanh tương tự như ở đậu tương (30) và lúa mì (27), nhưng nhiều hơn ở khoai tây (9), cà chua (21) gạo (15) và ngô (15). Kích thước bộ gen là 373 Mb đối với cây lanh, 1,1 Gb đối với đậu tương (Chu et al., 2021)và 16 Gb cho lúa mì (Z. Li et al., 2019) cho thấy không có mối quan hệ nào giữa kích thước bộ gen và số lượng thành viên họ gen PLATZ.
Sao chép gen là cơ chế chính thúc đẩy quá trình tiến hóa gen và hệ thống di truyền. Bộ gen thực vật có số lượng lớn các gen nhân đôi do các sự kiện nhân đôi cũ và các cặp gen nhân đôi hiện đại được bảo tồn tốt. Sự xuất hiện của các chức năng mới được tạo điều kiện thuận lợi bởi những bản sao này (Moore & Purugganan, 2003) (Panchy, Lehti-Shiu, & Shiu, 2016). Các gen FtPLATZ được sao chép song songvà các gen FtPLATZ được sao chép theo từng đoạn đã được báo cáo ở kiều mạch Tartary, chiếm 50% số gen FtPLATZ (7 trong số 14 gen). Do đó, nguyên nhân chính của việc khuếch đại gen FtPLATZ là do sao chép gen (Jing Li et al., 2022). Sự lặp lại song song và phân đoạn cũng đã được tìm thấy ở cà chua, cho thấy rằng các gen SlPLATZ có thể đã phân kỳ trong quá trình tiến hóa (Wai et al., 2020). Chúng tôi đã phát hiện 27 cặp gen sao chép đoạn trong số các gen LuPLATZ (Hình 4a), cho thấy rằng động lực chính của quá trình tiến hóa LuPLATZ là sự sao chép bộ gen. Ngoài ra, phân tích liên kết cho thấy số lượng cặp gen LuPLATZ tương đồng với cây hai lá mầm Arabidopsis nhiều hơn so với cây lúa một lá mầm, cho thấy mối quan hệ tiến hóa giữa gen LuPLATZ và AtPLATZ gần gũi hơn.
TÀI LIỆU THAM KHẢO
1. Azim, J. B., Khan, M. F. H., Hassan, L., Robin, A. H. K. J. P. b., & biotechnology. (2020). Genome-wide characterization and expression profiling of plant-specific PLATZ transcription factor family genes in Brassica rapa L. 8(1), 28-45.
2. Barkoula, N., Garkhail, S., Peijs, T. J. I. C., & Products. (2010). Biodegradable composites based on flax/polyhydroxybutyrate and its copolymer with hydroxyvalerate. 31(1), 34-42.
3. Cheng, F., Liu, S., Wu, J., & Fang, L. J. B. P. B. Sun Set al (2011) BRAD, the genetics and genomics database for Brassica plants. 11, 136.
4. Chu, J. S.-C., Peng, B., Tang, K., Yi, X., Zhou, H., Wang, H., . . . Feng, X. J. S. D. (2021). Eight soybean reference genome resources from varying latitudes and agronomic traits. 8(1), 164.
5. Fu, Y., Cheng, M., Li, M., Guo, X., Wu, Y., & Wang, J. J. I. j. o. m. s. (2020). Identification and characterization of PLATZ transcription factors in wheat. 21(23), 8934.
6. González-Morales, S. I., Chávez-Montes, R. A., Hayano-Kanashiro, C., Alejo-Jacuinde, G., Rico-Cambron, T. Y., de Folter, S., & Herrera-Estrella, L. J. P. o. t. N. A. o. S. (2016). Regulatory network analysis reveals novel regulators of seed desiccation tolerance in Arabidopsis thaliana. 113(35), E5232-E5241.
7. Goyal, A., Sharma, V., Upadhyay, N., Gill, S., & Sihag, M. (2014). Flax and flaxseed oil: an ancient medicine & modern functional food. J Food Sci Technol, 51(9), 1633-1653. doi:10.1007/s13197-013-1247-9
8. Han, X., Rong, H., Tian, Y., Qu, Y., Xu, M., & Xu, L.-a. J. F. i. P. S. (2022). Genome-wide identification of PLATZ transcription factors in Ginkgo biloba L. and their expression characteristics during seed development. 13, 946194.
9. Kim, J. H., Kim, J., Jun, S. E., Park, S., Timilsina, R., Kwon, D. S., . . . Nam, H. G. J. N. P. (2018). ORESARA15, a PLATZ transcription factor, mediates leaf growth and senescence in Arabidopsis. 220(2), 609-623.
10. Li, J., Feng, S., Zhang, Y., Xu, L., Luo, Y., Yuan, Y., . . . Feng, B. J. B. P. B. (2022). Genome-wide identification and expression analysis of the plant-specific PLATZ gene family in Tartary buckwheat (Fagopyrum tataricum). 22(1), 160.
11. Li, J., Zhao, Y., Zhang, Y., Ye, F., Hou, Z., Zhang, Y., . . . Tan, M. J. B. g. (2023). Genome-wide analysis of MdPLATZ genes and their expression during axillary bud outgrowth in apple (Malus domestica Borkh.). 24(1), 329.
12. Li, Z., Wang, M., Lin, K., Xie, Y., Guo, J., Ye, L., . . . Tong, Y. J. G. b. (2019). The bread wheat epigenomic map reveals distinct chromatin architectural and evolutionary features of functional genetic elements. 20, 1-16.
13. Liu, Y., Huang, Y., Li, Z., Feng, M., Ge, W., Zhong, C., & Xue, R. (2023). Genome-wide identification of the TGA genes in common bean (Phaseolus vulgaris) and revealing their functions in response to Fusarium oxysporum f. sp. phaseoli infection. Front Genet, 14, 1137634. doi:10.3389/fgene.2023.1137634
14. Mahboob, Z., El Sawi, I., Zdero, R., Fawaz, Z., Bougherara, H. J. C. P. A. A. S., & Manufacturing. (2017). Tensile and compressive damaged response in Flax fibre reinforced epoxy composites. 92, 118-133.
15. Moore, R. C., & Purugganan, M. D. J. P. o. t. N. A. o. S. (2003). The early stages of duplicate gene evolution. 100(26), 15682-15687.
16. Panchy, N., Lehti-Shiu, M., & Shiu, S.-H. J. P. p. (2016). Evolution of gene duplication in plants. 171(4), 2294-2316.
17. Prasad, K. (2000). Antioxidant Activity of Secoisolariciresinol Diglucoside-derived Metabolites, Secoisolariciresinol, Enterodiol, and Enterolactone. Int J Angiol, 9(4), 220-225. doi:10.1007/bf01623898
18. Shim, Y. Y., Gui, B., Arnison, P. G., Wang, Y., Reaney, M. J. J. T. i. f. s., & technology. (2014). Flaxseed (Linum usitatissimum L.) bioactive compounds and peptide nomenclature: A review. 38(1), 5-20.
19. Sun, Y., Liu, Y., Liang, J., Luo, J., Yang, F., Feng, P., . . . Zhao, T. J. F. i. p. s. (2023). Identification of PLATZ genes in Malus and expression characteristics of MdPLATZs in response to drought and ABA stresses. 13, 1109784.
20. Wai, A. H., Waseem, M., Khan, A. M. M., Nath, U. K., Lee, D. J., Kim, S. T., . . . Chung, M. Y. J. G. (2020). Genome-wide identification and expression profiling of the PDI gene family reveals their probable involvement in abiotic stress tolerance in tomato (Solanum lycopersicum L.). 12(1), 23.
21. Wang, A., Hou, Q., Si, L., Huang, X., Luo, J., Lu, D., . . . Xie, Y. J. P. P. (2019). The PLATZ transcription factor GL6 affects grain length and number in rice. 180(4), 2077-2090.
22. Wang, J., Ji, C., Li, Q., Zhou, Y., & Wu, Y. (2018). Genome-wide analysis of the plant-specific PLATZ proteins in maize and identification of their general role in interaction with RNA polymerase III complex. BMC Plant Biol, 18(1), 221. doi:10.1186/s12870-018-1443-x
23. Xie, D., Dai, Z., Yang, Z., Tang, Q., Sun, J., Yang, X., . . . Su, J. (2018). Genomic variations and association study of agronomic traits in flax. BMC Genomics, 19(1), 512. doi:10.1186/s12864-018-4899-z
24. Zheng, Y., Jiao, C., Sun, H., Rosli, H. G., Pombo, M. A., Zhang, P., . . . Giovannoni, J. J. J. M. p. (2016). iTAK: a program for genome-wide prediction and classification of plant transcription factors, transcriptional regulators, and protein kinases. 9(12), 1667-1670.